亚硫酸盐定序
编辑亚硫酸盐定序(也称为亚硫酸氢盐测序)是在常规测序之前使用亚硫酸氢盐处理 DNA 以确定甲基化模式。 DNA 甲基化是第一个发现的表观遗传标记,并且仍然是研究最多的。 在动物中,它主要涉及将甲基添加到二核苷酸 CpG 胞嘧啶残基的碳 5 位,并与转录活性的抑制有关。
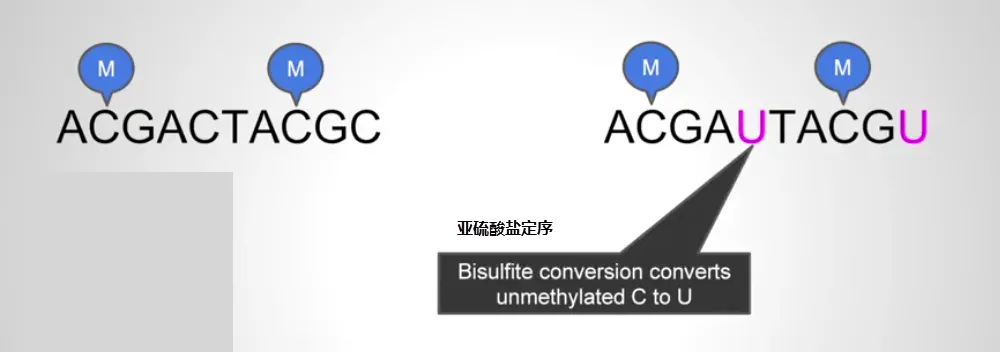
用亚硫酸氢盐处理 DNA 会将胞嘧啶残基转化为尿嘧啶,但不会影响 5-甲基胞嘧啶残基。 因此,用亚硫酸氢盐处理过的 DNA 仅保留甲基化胞嘧啶。 因此,亚硫酸氢盐处理会根据单个胞嘧啶残基的甲基化状态在 DNA 序列中引入特定变化,从而产生有关 DNA 片段甲基化状态的单核苷酸分辨率信息。 可以对改变的序列进行各种分析以检索此信息。 因此,该分析的目的简化为区分亚硫酸氢盐转化产生的单核苷酸多态性。
方法
编辑亚硫酸盐定序对亚硫酸氢盐处理的基因组 DNA 应用常规测序方法,以确定 CpG 二核苷酸的甲基化状态。 还采用其他非测序策略来研究特定位点或全基因组水平的甲基化。 所有策略都假设亚硫酸氢盐诱导的未甲基化胞嘧啶向尿嘧啶的转化是完整的,并且这是所有后续技术的基础。 理想情况下,使用的方法将分别确定每个等位基因的甲基化状态。 亚硫酸氢盐测序的替代方法包括联合亚硫酸氢盐限制性分析和甲基化 DNA 免疫沉淀 (MeDIP)。
分析亚硫酸氢盐处理的 DNA 的方法正在不断发展。 为了总结这些快速发展的方法,已经撰写了大量评论文章。
这些方法通常可分为基于甲基化特异性 PCR (MSP) 的策略和采用在非甲基化特异性条件下进行的聚合酶链反应(PCR)的策略。 基于微阵列的方法也使用基于非甲基化特异性条件的 PCR。
基于非甲基化特异性 PCR 的方法
编辑直接测序
第一个报道的使用亚硫酸氢盐处理的 DNA 的甲基化分析方法利用 PCR 和标准双脱氧核苷酸 DNA 测序来直接确定耐亚硫酸氢盐转化的核苷酸。 引物被设计为链特异性和亚硫酸氢盐特异性(即含有非 CpG 胞嘧啶的引物,因此它们与非亚硫酸氢盐处理的 DNA 不互补),侧翼(但不涉及)感兴趣的甲基化位点。 因此,与甲基化特异性 PCR 不同,它会同时扩增甲基化和非甲基化序列。 未甲基化胞嘧啶的所有位点在所得的有义链扩增序列中显示为胸腺嘧啶,在扩增的反义链中显示为腺嘌呤。 通过将高通量测序接头整合到 PCR 引物中,可以对 PCR 产物进行大规模平行测序。 或者,可以对 PCR 产物进行克隆和测序,这需要大量的劳动。 巢式 PCR 方法可用于增强测序产物。
使用亚硫酸氢盐处理的 DNA 的所有后续 DNA 甲基化分析技术均基于 Frommer 等人的这份报告。 虽然大多数其他模式不是真正的基于测序的技术,但术语亚硫酸氢盐测序通常用于描述一般的亚硫酸氢盐转化 DNA 甲基化分析技术。
焦磷酸测序
焦磷酸测序也被用于分析亚硫酸氢盐处理的 DNA,而不使用甲基化特异性 PCR。 在对感兴趣的区域进行 PCR 扩增后,焦磷酸测序用于确定该区域中特定 CpG 位点的亚硫酸氢盐转化序列。 可以根据序列延伸过程中 C 和 T 的掺入量定量确定各个位点的 C 与 T 的比率。 这种方法的主要限制是技术成本。 然而,焦磷酸测序确实允许扩展到高通量筛选方法。
Wong 等人最近描述了对该技术的进一步改进,它使用等位基因特异性引物,将单核苷酸多态性整合到测序引物的序列中,从而允许分别分析母本和父本等位基因。 该技术对于基因组印迹分析特别有用。
甲基化敏感性单链构象分析(MS-SSCA)
该方法基于为单核苷酸多态性 (SNP) 分析开发的单链构象多态性分析 (SSCA) 方法。
内容由匿名用户提供,本内容不代表vibaike.com立场,内容投诉举报请联系vibaike.com客服。如若转载,请注明出处:https://vibaike.com/229253/
