DNA微阵列
编辑DNA 微阵列是附着在固体表面的微观 DNA 点的集合。 科学家使用 DNA 微阵列同时测量大量基因的表达水平或对基因组的多个区域进行基因分型。 每个 DNA 点包含特定 DNA 序列的皮摩尔(10-12 摩尔),称为探针(或记者或寡核苷酸)。 这些可以是用于在高严格条件下杂交 CDNA 或 cRNA(也称为反义 RNA)样品(称为目标)的基因或其他 DNA 元件的一小部分。 探针-目标杂交通常通过检测荧光团、银或化学发光标记的目标来检测和量化,以确定目标中核酸序列的相对丰度。
原则
编辑微阵列背后的核心原理是两条 DNA 链之间的杂交,即互补核酸序列通过在互补核苷酸碱基对之间形成氢键而相互特异性配对的特性。 核苷酸序列中的大量互补碱基对意味着两条链之间更紧密的非共价键合。 洗掉非特异性键合序列后,只有强配对的链会保持杂交状态。 与探针序列结合的荧光标记的靶序列产生的信号取决于杂交条件(例如温度)和杂交后的洗涤。 来自一个点(特征)的信号的总强度取决于与该点上存在的探针结合的目标样本的量。 微阵列使用相对定量,其中将特征的强度与不同条件下的相同特征的强度进行比较,并且特征的身份通过其位置已知。
用途和类型
编辑存在许多类型的阵列,最广泛的区别在于它们是在空间上排列在表面上还是编码珠上:
- 传统的固相阵列是一组有序的微观点,称为特征,每个点都有数千个相同且特定的探针附着在固体表面,例如玻璃、塑料或硅生物芯片(通常称为基因组芯片, DNA 芯片或基因阵列)。 数以千计的这些特征可以放置在单个 DNA 微阵列上的已知位置。
- 另一种微珠阵列是聚苯乙烯微珠的集合,每个微珠都有一个特定的探针和两种或多种染料的比例,不会干扰目标序列上使用的荧光染料。
DNA 微阵列可用于检测 DNA(如比较基因组杂交),或检测可能会或可能不会翻译成蛋白质的 RNA(最常见的是逆转录后的 cDNA)。 通过 cDNA 测量基因表达的过程称为表达分析或表达谱分析。
应用包括:
针对特定作物量身定制的专用阵列在分子育种应用中越来越受欢迎。 将来,它们可用于在早期阶段筛选幼苗,以减少在育种操作中试验的不需要的幼苗数量。
制造
微阵列可以用不同的方式制造,这取决于被检查的探针数量、成本、定制要求和所提出的科学问题的类型。 来自商业供应商的阵列可能只有 10 个探针或多达 500 万个或更多的微米级探针。
点阵与原位合成阵列
可以使用多种技术制造微阵列,包括在载玻片上使用尖头针印刷、使用预制掩模的光刻、使用动态微镜设备的光刻、喷墨印刷或微电极阵列上的电化学。
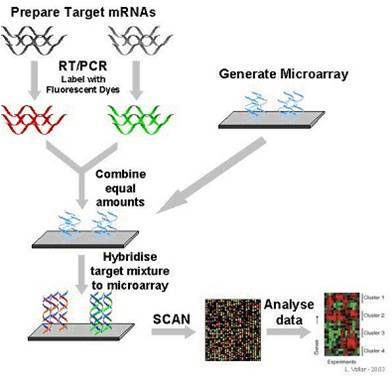
在斑点微阵列中,探针是寡核苷酸、cDNA 或与 MRNA 相对应的 PCR 产物的小片段。 探针在沉积在阵列表面之前合成,然后点在玻璃上。 一种常见的方法是利用由机械臂控制的一系列细针或针,机械臂浸入含有 DNA 探针的孔中,然后将每个探针放置在阵列表面的指定位置。 由此产生的探针网格代表准备好的探针的核酸图谱,并准备好接收来自实验或临床样本的互补 cDNA 或 cRNA 靶标。世界各地的研究科学家使用这种技术在他们的内部生产打印微阵列 自己的实验室。
内容由匿名用户提供,本内容不代表vibaike.com立场,内容投诉举报请联系vibaike.com客服。如若转载,请注明出处:https://vibaike.com/215351/
